Ausbruchsaufklärung mit Eigenkontrollisolaten – Nutzen einer hochauflösenden Typisierung
Sabine Horlacher, Ekkehard Hiller, Matthias Contzen
Mittels Next Generation Sequencing (NGS) können genetische Eigenschaften in einzelnen Bakterien, aber auch Verwandtschaftsverhältnisse zwischen unterschiedlichen Bakterien-Isolaten bestimmt werden. Auf Initiative des Ministeriums für Ernährung, Ländlichen Raum und Verbraucherschutz (MLR) werden in Baden-Württemberg seit einigen Jahren Isolate von krankheitserregenden Mikroorganismen, die im Rahmen von betrieblichen Eigenkontrolluntersuchungen gewonnen wurden, am CVUA Stuttgart gesammelt und für weitere Analysen genutzt. Diese umfassen Programme zur Untersuchungen im Zusammenhang mit epidemiologischen Ermittlungen bei Humanerkrankungen aber auch die Untersuchung auf mehrfach resistente Erreger.
Als Zentrallabor für Lebensmittel im Erkrankungszusammenhang in Baden-Württemberg ist das Chemische und Veterinäruntersuchungsamt Stuttgart (CVUA Stuttgart) auch in die epidemiologischen Ermittlungen bei Humanerkrankungen eingebunden. Neben Isolaten von pathogenen Mikroorganismen aus Lebensmitteln und Umgebungsproben, die im Zusammenhang mit Erkrankungsfällen beim Menschen von der Lebensmittelüberwachungsbehörde (LMÜ) erhoben werden, nutzen wir auch Isolate aus betrieblichen Eigenkontrollen. Lebensmittelunternehmer überprüfen hierbei ihre Produktion i. d. R. in selbst festgelegten Abständen und Umfängen auf ihre Lebensmittelsicherheit. Sofern bei diesen Eigenkontrolluntersuchungen pathogene Mikroorganismen wie Salmonellen oder Listerien nachgewiesen werden, sind die Lebensmittelunternehmer bzw. das von ihnen beauftragte Labor zur Meldung an die in ihrem Stadt- oder Landkreis zuständige LMÜ verpflichtet.
Generelle Anforderung der Isolate
Seit mehreren Jahren werden diese Angaben in Baden-Württemberg auf Veranlassung des MLR systematisch genutzt, um Isolate dieser nachgewiesenen Mikroorganismen anzufordern und weiteren Untersuchungen wie z. B. eine Feintypisierung oder eben epidemiologische Abgleiche mit Erkrankungsfällen beim Menschen durchzuführen. Als Rechtsgrundlage dient § 3 der Zoonose-Verordnung [1].
Danach sind Mikroorganismen-Kulturen, die im Rahmen der Labordiagnostik z. B. auf Agarplatten gewonnen werden können, der zuständigen Behörde auf Verlangen auszuhändigen. In Baden-Württemberg ist die Abt. Lebensmittelmikrobiologie des CVUA Stuttgart zentral für die Entgegennahme und weitere Bearbeitung derartiger Isolate zuständig.
Infokasten
Was gilt als Eigenkontrollen? (§ 3 Abs. 1 Zoonose-VO)
- Kontrollen nach Art. 3 (1) der VO (EG) Nr. 2073/2005 (…) oder anderen betriebseigenen Kontrollen
- alle Untersuchungen von Lebensmitteln (z. B. Rohwaren, Zwischenprodukte, fertige Produkte) auf Zoonoseerreger
- Untersuchungen auf Listeria monocytogenes von Produktresten von Lebensmitteln bei der Herstellung und Bearbeitung von verzehrfertigen Lebensmitteln
- Untersuchungen von Schmierwasser auf Listeria monocytogenes in der Herstellung von verzehrfertigem Käse
- Untersuchungen auf Listeria monocytogenes zur Prüfung des Reinigungs- und Desinfektionserfolgs in Betriebseinrichtungen
In den letzten Jahren erhalten wir eine jährlich zunehmende Anzahl von pathogenen Keimen aus verschiedenen Lebensmittelprodukten oder der Umgebungsbeprobung der Herstellerbetriebe (Abb. 1). Im Jahre 2022 betrug der Umfang der Einsendungen 172 Kulturen, die auf Agarplatte, in Kryoröhrchen oder mittels Tupfer eingesendet wurden.
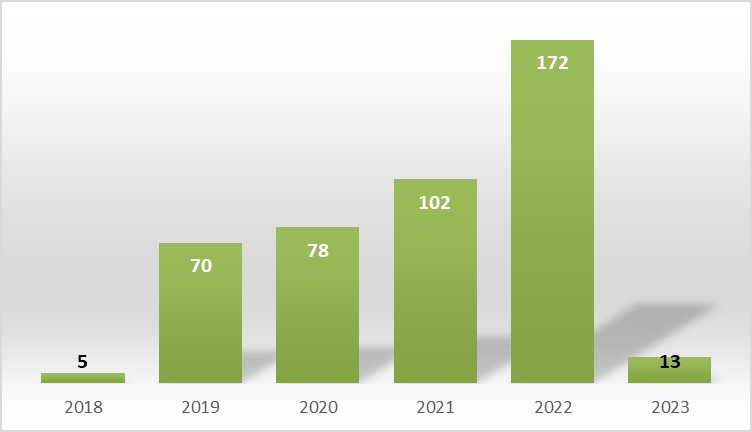
Abbildung 1: Anzahl eingesendeter Isolate (Stand: Feb. 2023)
Im Laufe der Jahre sind so insbesondere 290 Listeria monocytogenes und 87 Salmonellen zusammengetragen worden (Abb. 2). Vereinzelt erhalten wir auch Campylobacter-Isolate. Die eingesandten Kulturen werden im Labor des CVUA Stuttgart auf Reinheit überprüft sowie eine Speziesdifferenzierung mittels MALDI-TOF MS, FT-IR bzw. bei Salmonellen auch durch Agglutination durchgeführt.
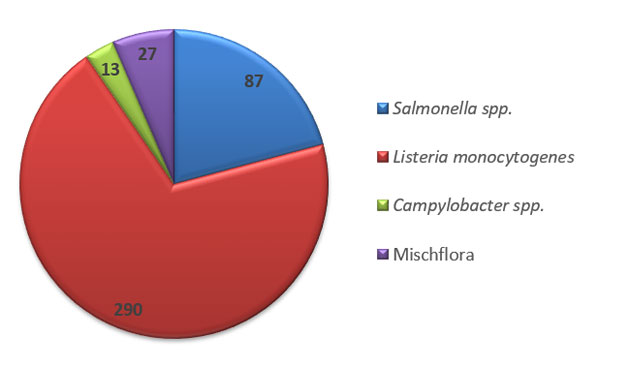
Abbildung 2: Verteilung der Mikroorganismen (Stand: Feb. 2023)
Außerdem werden die Isolate mit einer Ganzgenomsequenzierung (WGS) analysiert, die per Next Generation Sequencing (NGS) durchgeführt wird. Anschließend werden die so gewonnenen Sequenzen mit denen anderer Lebensmittel-Isolate aus ganz Baden-Württemberg und Isolaten aus Humanerkrankungsfällen innerhalb des Landes sowie mit anderen Bundesländern abgeglichen. Hierzu werden gemeinsam mit den anderen CVUAs in Baden-Württemberg entsprechende Plattformen wie z. B. miGenomeSurv [2] oder das WGS Portal der Europäischen Behörde für Lebensmittelsicherheit (EFSA) genutzt.
Sofern sich aus diesen Abgleichanalysen relevante Übereinstimmungen zwischen Lebensmittelkeimen und aus Patienten isolierten Bakterien ergeben, werden das MLR und die zuständige LMÜ darüber informiert, um ihrerseits Maßnahmen wie z. B. Rückverfolgung oder Prüfung der Plausibilität in Bezug auf die Epidemiologie einzuleiten.
Nutzen aus der Datenanalyse
Die Ergebnisse dieser Analysen können vielfältig sein. So wurde z. B. in einem Lebensmittel-Herstellerbetrieb (Betrieb A) das Vorhandensein eines sogenannten Hauskeimes festgestellt, der sich trotz aller Reinigungs- und Desinfektionsmaßnahmen in der Produktion festgesetzt und über mehrere Jahre hinweg immer wieder in Erzeugnissen dieses Herstellers nachverfolgbar zu finden war (Abb. 3).
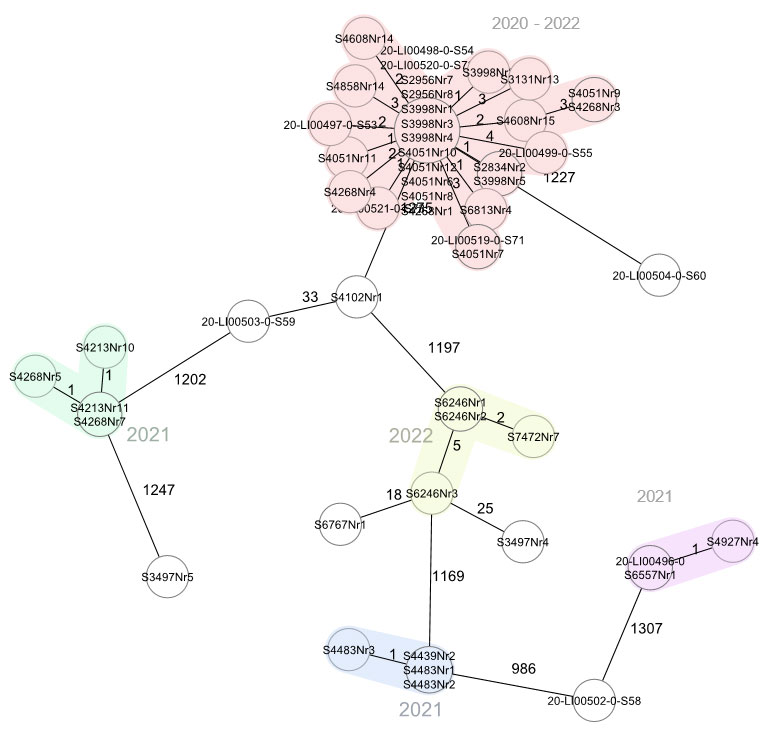
Abbildung 3: Minimum Spanning Tree (Ridom SeqSphere+) aller Isolate des Betriebs A ab 2020. Die Zahlen zwischen den Sequenzdatensätzen bezeichnen die Anzahl unterschiedlicher Allele (Alleldifferenzen; AD), also den „genetischen Abstand“. Farbig hinterlegte Einträge gehören einem Cluster an, also einer Gruppe von genetisch sehr ähnlichen Isolaten.
In anderen Fällen zeigten die Untersuchungen im Gegensatz dazu beispielsweise, dass ein Eintrag von Listeria monocytogenes in Betrieb B immer wieder über die zugelieferten Rohstoffe erfolgte (Abb. 4).
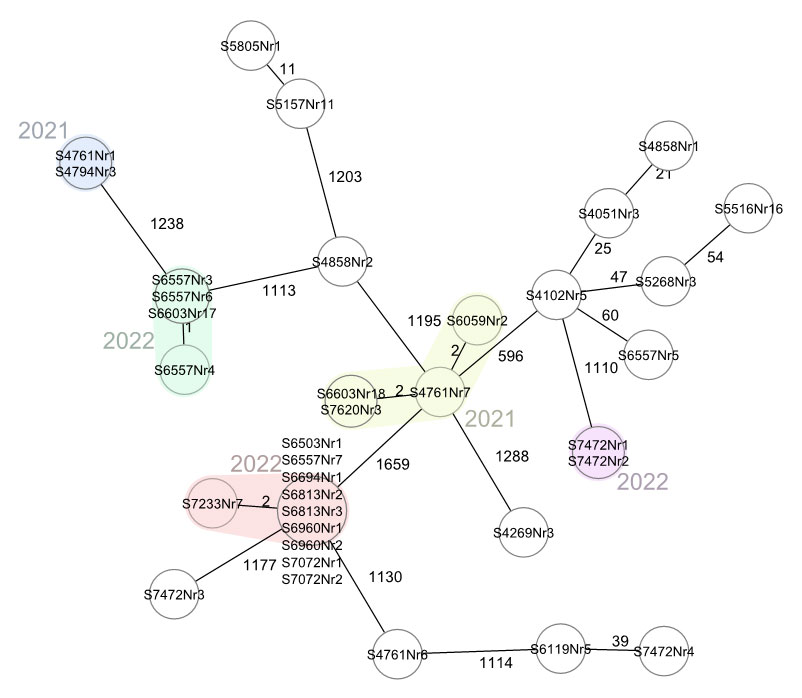
Abbildung 4: Minimum Spanning Tree (Ridom SeqSphere+) aller Isolate des Betriebs B ab 2021
Die in diesen beiden Szenarien (seitens LMÜ wie auch seitens des Herstellers) zu treffenden Maßnahmen unterscheiden sich hier grundlegend.
Durch den Abgleich mit Daten von Patientenkeimen, die beispielsweise das Landesgesundheitsamt oder das Robert-Koch-Institut zur Verfügung stellen, wurde ebenfalls bereits in mehreren Fällen ein bisher unentdeckt gebliebener möglicher Zusammenhang mit Lebensmitteln entdeckt (Abb. 5). Wichtig ist hierbei jedoch, dass die WGS-Ergebnisse immer nur einen ersten Hinweis auf einen epidemiologischen Zusammenhang darstellen. Dieser muss durch nachfolgende Untersuchungen (z. B. Prüfung hinsichtlich der Lieferkette oder Abgleich mit den tatsächlich von erkrankten Personen verzehrten Lebensmitteln) bestätigt werden.
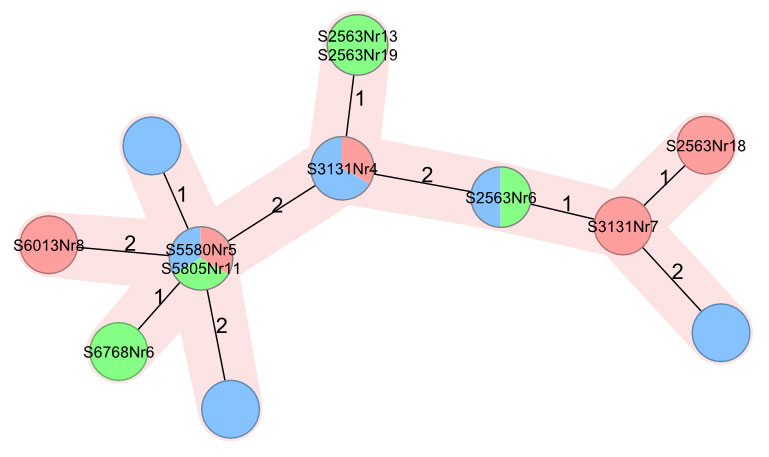
Abbildung 5: Minimum Spanning Tree (Ridom SeqSphere+) von Isolaten eines Ausbruches. Humanisolate sind blau gefärbt, Eigenkontrollisolate grün und amtliche Planproben rot.
NGS als neue Technik ermöglicht es so Zusammenhänge aufzudecken, die bisher vollständig unbemerkt geblieben sind, oder erst nach langwierigen epidemiologischen Untersuchungen feststellbar waren. So können kontaminierte Lebensmittel schneller identifiziert und Erkrankungsausbrüche beim Menschen gestoppt werden.
Diese Weiterentwicklung der epidemiologischen Methoden wird somit durch die konsequente Nutzung der bestehenden Rechtsgrundlagen insbesondere durch Anforderung der Eigenkontrollisolate in Baden-Württemberg seit mehreren Jahren effektiv zum Schutz der Verbraucher eingesetzt.
Infokasten
Einsendung der Isolate ans CVUA Stuttgart
Angabe folgender Informationen:
- Art des Isolates (z. B. Salmonelle)
- Untersuchte Matrix (Lebensmittel, Umgebung)
- Untersuchungsdatum
- entnommen bei: Betrieb, Ort / bei Handelsware: Herstellerbetrieb, Ort
- Interne Privatlabor-Nr. des Isolates und Privatlabor
Einsendung als Platte/Röhrchen/Tupfer an:
CVUA Stuttgart, Lebensmittelmikrobiologie
(z. Hd. Fr. Dr. Horlacher)
Schaflandstr. 3/2, 70736 Fellbach
Quellen
[1] Zoonose-VO: Verordnung mit lebensmittelrechtlichen Vorschriften zur Überwachung von Zoonosen und Zoonoseerregern vom 8. August 2007 (BGBl. I S. 1816, 1871), die zuletzt durch Artikel 1 der Verordnung vom 19. Juni 2020 (BGBl. I S. 1480) geändert worden ist.
[2] miGenomeSurv zuletzt aufgerufen am 06.03.2023